近日,一项基因组多维解析技术全国重点实验室和深圳华大生命科学研究院的研究成果登上《基因组生物学》(Genome Biology)。团队研发了一款名为SpaSEG的AI“解码器”。这一工具能够处理多项核心任务,包括识别组织空间结构域、检测空间高变基因、整合多张组织切片以及解析细胞间的相互作用,为空间转录组分析提供功能强大的统一框架。

论文成果截图,图源:《基因组生物学》
想象一下,如果我们想了解一座城市的运作模式,只拿到一份所有建筑物的清单,是不是很难理解商业区、住宅区和工业区是如何协同工作得?但如果有一张高清卫星地图,所有功能区一目了然,城市的秘密便豁然开朗。
在生命科学研究中,科学家们也面临着类似的挑战。基因测序就像那份“建筑物清单”,它告诉我们组织里有哪些细胞和基因,却丢失了它们在组织内的“空间地址”。空间转录组学技术,如同为生命组织绘制“高清卫星地图”,它能同时捕捉基因信息和其空间位置,为我们揭示细胞是如何“安家落户”、相互“沟通”的,从而推动我们对发育、衰老和疾病(尤其是癌症)的理解。
然而,如何从这些复杂如星空图谱的数据中,快速、准确地解读出有价值的生物学信息,一直是该领域的核心挑战,尤其是在面对不同技术平台产生的不同形式的数据以及庞大的数据规模时。这项成果正是要解决这一难题。
简单来说,SpaSEG就像是一个训练有素的“AI分析师”。它能够根据复杂的空间转录组数据,自动识别出组织中的功能分区(如不同结构或病灶区)、找到基因表达存在空间差异的区域、拼接多个组织切片,并推断细胞之间的通信模式。
研究团队介绍,这款模型的独特之处在于,它首次将空间转录组数据“图像化”处理,借助卷积神经网络(CNN)——一种常用于图像识别的AI技术——对组织结构进行“看图识区”。这一方法不仅更智能,而且具有更强的适应性和处理速度,突破了传统工具在兼容性、精度和运算效率上的瓶颈。
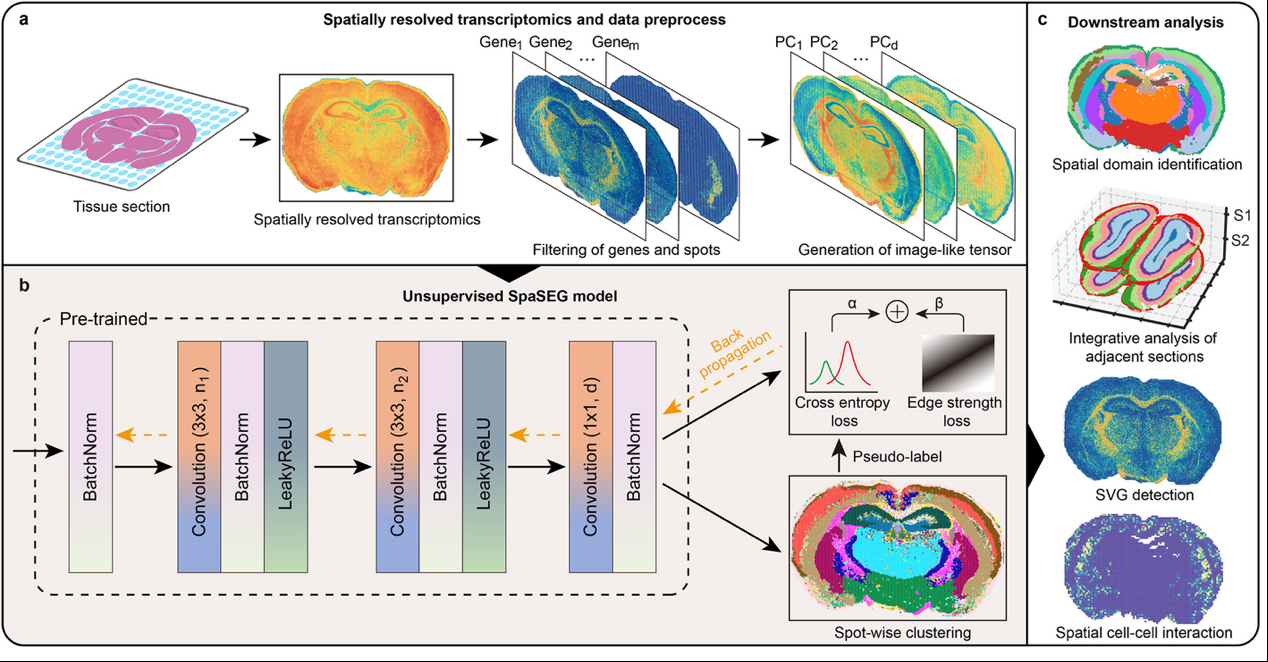
SpaSEG模型架构及多任务下游分析,图源:《基因组生物学》
SpaSEG最突出的优势之一,是其显著的跨平台兼容性和处理多尺度分辨率数据的能力。实验表明,SpaSEG在面向Stereo-seq(覆盖从Bin200到Bin20的多种分辨率)等多种平台不同分辨率的空间转录组数据时,识别空间结构域时的准确性均显著优于现有方法。
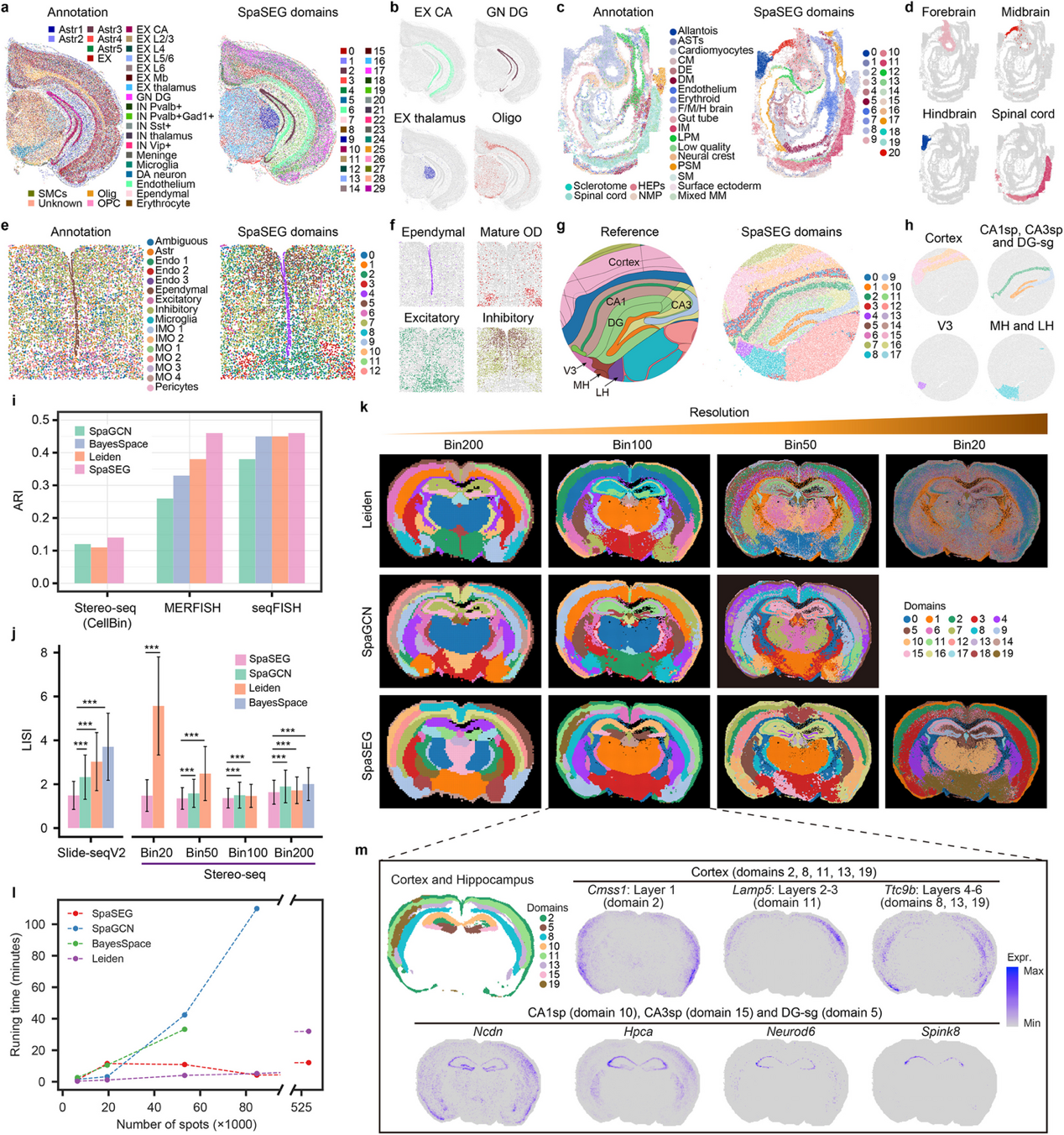
跨平台多尺度分辨率的组织空间结构域识别,图源:《基因组生物学》
SpaSEG通过精准对齐相邻组织切片的空间坐标和基因表达模式实现了跨切片空间结构域对齐,从而支持3D组织结构重建。在小鼠嗅球Stereo-seq数据中,SpaSEG成功对齐了多个解剖结构,对齐精度提升了20%-30%,且无需依赖复杂的对齐算法。
基因组多维解析技术全国重点实验室、深圳华大生命科学研究院的白勇副研究员、刘传宇研究员和金鑫研究员为论文共同通讯作者。白勇、深圳华大生命科学研究院郭翔宇和刘可茵为论文共同第一作者。本项目获得国家重点研发计划资助。本研究使用的数据均为已发表的公开数据集。本研究已通过伦理审查,严格遵循相应法规和伦理准则。
编辑 黄小菊 审读 郭建华 二审 周梦璇 三审 刘思敏